Os vírus da Gripe Suína (SIV; siglas em inglês) pertencem ao género Influenzavirus tipo A da familia Orthomyxoviridae. Estes vírus subclassificam-se com base nas glicoproteínas de superficie: a hemaglutinina (HA) e a neuraminidase (NA), das quais se descrevem 17 e 10 tipos, respectivamente. A combinação de um tipo de HA com um tipo de NA é o que define o que conhecemos como subtipo. Assim, os subtipos maioritários no porco são o H1N1, o H1N2 e o H3N2.
Com base nos diferentes estudos realizados na Europa, podemos afirmar que a seroprevalência dos três subtipos (H1N1, H1N2 e H3N2) é muito alta nos países com uma produção suína importante. Por exemplo, na Alemanha, Bélgica, Dinamarca ou Espanha, a seroprevalência dos três subtipos é superior a 30%. Concretamente em Espanha, 94% das explorações são positivas pelo menos a um subtipo. Em França ou no Reino Unido detectam-se elevadas prevalências de H1N1 ou H1N2; contudo, parece que o subtipo H3N2 não estaría presente pelo menos durante os últimos anos. Em países com uma produção menor, como por exemplo a República Checa, a Irlanda ou a Polónia, a prevalência dos SIV parece ser inferior.

Numa mesma exploração, é possível detectar de forma directa (mediante PCR) ou de forma indirecta (mediante inibição da hemaglutinação) mais de um subtipo de SIV circulante. Com efeito, esta situação foi demonstrada em explorações da Holanda, França ou Espanha, entre outros. Individualmente, deve-se acrescentar que a proporção de animais que apresentam anticorpos para mais de um subtipo pode chegar a ser elevada tanto em porcas reprodutoras como em porcos de engorda. Este facto sugere que a infecção por SIV é activa e frequente. Apesar disso, a presença do vírus nem sempre está associada a surtos de patologia respiratoria; com efeito, existe um elevado número de explorações positivas ao vírus sem clínica aparente. Em definitivo, estas descobertas sugerem que o SIV se apresenta de maneira importante como uma infecção enzoótica.
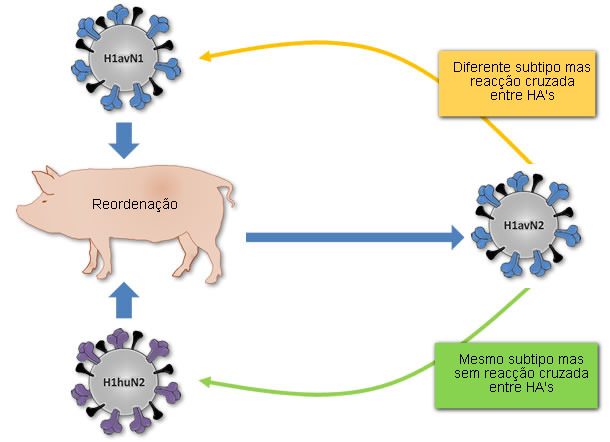
Figura 1. Origem do vírus H1avN2 descrito na Dinamarca e França. Este vírus gera-se a partir da reordenação dos vírus H1avN1 (que fornece a HA) e o vírus H1huN2 (que fornece a NA). Tal como o vírus H1huN2, o H1avN2 vírus pertence ao subtipo H1N2, mas a sua HA não reagirá de forma cruzada com este vírus, enquanto que lo fará com a HA do vírus H1avN1 ainda que este pertença a um subtipo diferente (H1N1).
A classificação por subtipos é útil para poder simplificar e entender a epidemiologia e diversidade dos vírus da gripe; contudo, o certo é que a diversidade dos SIV é mais complexa. Como muito bem resume a Dra. Simon noutro artigo publicado nesta mesma página web, na Europa podem-se distinguir, no mínimo, 4 linhagens principais em função do tipo de HA que contenham. Assim, distinguiríamos os relacionados com os H1N1 tipo aviar (H1avN1), os relacionados com os H1N2 (H1huN2), os relacionados com os H1N1 pandémico (H1N1pdm) e finalmente os relacionados com os H3N2.
É necessário recordar que os SIV, tal como qualquer outro vírus pertencente aos influenzavirus A, são compostos por um genoma segmentado em 8 fragmentos de RNA o qual permite que evoluam mediante dois mecanismos: 1) a deriva antigénica; mutações que quando se produzem em lugares específicos da HA (principalmente) ou na NA podem gerar variantes víricas que escapem da imunidade gerada previamente na população por vírus ancestrais (causa principal da gripe sazonal no ser humano), e 2) a reordenação genética, a qual se produz quando dois ou mais vírus trocam fragmentos de RNA (por exemplo, a geração no ano 1994 da estirpe H1huN2, resultante da reordenação entre um H1N1 humano e o H3N2 suíno).
Apesar de que os 3 H1 distintos (H1avN1, H1huN2, e H1N1pdm) tenham tido um ancestral comum nalgum momento, não cruzam a nível antigénico ou fazem-no de forma limitada. Provavelmente, isto se deva a que as três H1 evoluiram por deriva antigénica de forma isolada em três espécies distintas antes de se introduzirem no porco na Europa; em aves o H1avN1, em humanos o H1huN2 e em porcos na America do Norte o H1N1pdm. Por outro lado, a reordenação genética pode gerar novas estirpes de vírus da gripe que, ainda que pertencendo a um subtipo, podem cruzar antigenicamente com vírus pertencentes a outros subtipos. Um claro exemplo disso é a estirpe H1avN2 descrita na Dinamarca e em França. A referida estirpe contém uma HA relacionada com a presente nas estirpes H1avN1 e por consequência não cruza antigenicamente com a estirpe H1huN2, mas sim que o faz com as estirpes H1avN1.
A diversidade genética e antigénica dos vírus da gripe deve entender-se como algo dinâmico e em evolução constante. Os trabalhos realizados na Alemanha, Dinamarca, Espanha ou Itália, recentemente publicados, assim o demonstram. Nesses trabalhos isolaram-se novas estirpes produto da reordenação entre as estirpes de SIV suínas e estirpes da gripe sazonal humana. Por todas estas razões, é de vital importância manter e, inclusive fomentar, uma vigilância epidemiológica activa do SIV com o objectivo de conhecer as estirpes presentes no efectivo suinícola bem como a sua prevalência e impacto.



