Ainda que a PRRS seja uma doença relativamente nova, que apareceu pela primeira vez no Canadá em 1979, actualmente é endémica em quase todos os países do mundo e causa perdas económicas devastadoras devido à redução da produção de leitões, problemas de reprodução e transtornos respiratórios. Vários cálculos sugerem que entre um quarto e um terço do custo atribuído a doenças infecciosas deve-se ao PRRS. Frequentemente a doença é persistente, de maneira que o animal recupera-se mas continua a infectar os porcos não expostos. Isto sugere que pode haver uma produção tardia de anticorpos neutralizantes, uma redução da resposta imunitária tanto inata como adquirida e uma mutação alta do vírus PRRS durante a replicação, aumentando a sua diversidade genética.
Uma colaboração internacional coordenada está a investigar vários aspectos sobre a PRRS, mas o “santo graal” de produzir porcos geneticamente resistentes está a ser bastante difícil. No entanto as ferramentas e tecnologias desenvolvidas nos projectos ‘genoma’ estão a fornecer novas oportunidades para destrinçar os componentes genéticos da resposta à infecção de PRRS.

Existem muitas evidências de uma variação significativa da resposta e da tolerância entre raças. Por exemplo, estudos alemães mostraram que, após uma exposição a PRRS, uma linha de porco miniatura mostrou uma duração reduzida da virémia e persistência e também uma carga viral consideravelmente mais baixa que uma linha comercial de Pietrain. Nos EUA, foram demonstradas diferenças muito significativas na resposta de porcos Duroc-Hampshire em relação a Large White-Landrace.
Um estudo colaborativo recente do Instituto Roslin de Edimburgo foi o primeiro estudo SNP de todo o genoma em que se perguntou “pode ser detectada uma parte da variação genética por marcadores genéticos?” Os investigadores analisaram dados acumulados de mais de 8000 ninhadas numa só exploração por um periodo de 10 anos, durante o qual foram reportados dois surtos de PRRS. Através de um chip SNP de 7K identificaram-se vários SNP significantes a actuar independentemente, o que sugere que há muitas regiões genéticas que regulam a tolerância ao PRRS. Os genótipos incluem Landrace, Large White, Pietrain, Meishan, Duroc e vários cruzamentos. Curiosamente, o impacto do vírus PRRS foi maior na raça Meishan que nos seus equivalentes europeus. Noutras explorações está a ser realizado um estudo utilizando um chip SNP maior e mais actual para ver se os SNP identificados continuam a ser significativos entre explorações e também para verificar se há alguns efeitos adversos correlacionados com as características produtivas.
Entretanto, na Itália, foi compilada uma base de dados que combina amostras de ADN com dados genealógicos e de produtividade com dados sanitários produtivos. Para tal foram utilizados mais de 5000 animais de quatros genótipos (Duroc, Landrace, Large White e Pietrain) de 18 explorações geneticamente relacionadas. Foram identificados vários SNP muito significativos, e o trabalho continua a investigar vários marcadores e se estes são consistentes ao longo dos testes.
Representação na escala multidimensional
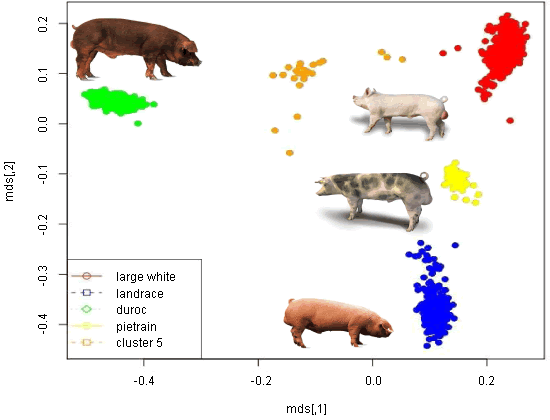
Relação genómica – Escala multidimensional (MDS): distribuição de clusters SNP por raças.
Os resultados das investigações até ao momento sugerem que há muitas variações genéticas na resposta à infecção para esperar melhorias na resistência ou na tolerância pela selecção, pelo que há razões para acreditar que seria possível produzir animais comerciais com maior resistência ou tolerância à doença. No entanto, a complexidade desta doença e o alto número de marcadores potencialmente envolvidos sugere que a rentabilidade da produção destes animais em grandes quantidades não poderá ser possível até dentro de uns anos.
Definições:
Um chip SNP é um pequeno pedaço de silicone que contém muitas "sondas" de ADN. Estas aderem muito selectivamente às moléculas de ADN cujas sequências de aminoácidos são complementares - a cada T corresponde uma A e a cada G uma C. Podem ser utilizados para identificar a presença de sequências específicas de DNA e procurar sequências de ADN que diferem em "polimorfismos simples".
SNP (pronunciada SNIP) quer dizer "polimorfismos simples". É a diferença só de um aminoácido entre individuos ou sequência de ADN. Os SNP são as variações genéticas mais comuns.
Livestock Genetics Ltd. Reino Unido



